
1. 前言
质量控制是在测序信息的分析开始之前的尤为重要的一步,因为后期的分析都是建立在这些数据的基础上的,所以前期数据的好坏直接影响了后期分析结果的质量高低、优良,因此做好质量控制非常重要。在二代测序中,测出来的片段都不长,而且还有一些PCR使用的接头也在其中,这些都会影响到后面的分析,因此我们要做的就是先把接头去掉,然后再筛选掉比较短的序列,比如使用Trinity进行拼接时,kmer的长度最小为50bp,小于这个阈值就无法拼接,因此在这个基础上,我们可以开始做质量控制了。
二代测序数据的特点:大量的短序列(150-250bp)、双末端测序、末端质量较低。因此,在利用我们的测序数据进行分析之前,首先需要过滤掉低质量的碱基与序列,以确保分析结果的准确性。
2. 二代测序一般质控步骤
切除尾端碱基质量小于指定值(一般为20)的碱基。可以简单的单碱基修剪,也即从末端开始进行删除,直到读取碱基质量高于20;也可以进行滑窗修剪,也即从末端开始以指定碱基数目的滑窗开始修剪,直到滑窗内碱基平均质量高于20。
去除末端修剪后长度小于指定值的reads。不同项目指定值不同,一般宏基因组去掉小于50bp的reads(50bp已不够产生k-mer),而扩增子测序则根据raw reads长度和PCR插入片段的长度来确定,例如V4区大概260bp,那么可以去掉双末端reads之和小于280bp的(否则不足以拼接)。
其他一些要求,例如去除含有N(即无法读取位点)过多的reads、去除完全重复的reads等。
3. trimmomatic介绍
trimmomatic是一款用来处理illumina测序数据的工具,可以是单条的single reads,也可以是成对的pairend reads。支持压缩格式数据。功能和其他数据处理的程序都差不多,主要包括,
- 去除adapter序列以及测序中其他特殊序列;
- 采用滑动窗口的方法,切除或者删除低质量碱基
- 去除头部低质量以及N碱基过多的reads;
- 去除尾部低质量以及N碱基过多的reads;
- 截取固定长度的reads;
- 丢掉小于一定长度的reads;
- Phred 质量值转换
Trimmomatic 发表的文章至今已被引用了 2810 次,是一个广受欢迎的 Illumina 平台数据过滤工具。其他平台的数据例如 Iron torrent ,PGM 测序数据可以用 fastx_toolkit 、NGSQC toolkit 来过滤。
Trimmomatic 支持多线程,处理数据速度快,主要用来去除 Illumina 平台的 Fastq 序列中的接头,并根据碱基质量值对 Fastq 进行修剪。软件有两种过滤模式,分别对应 SE 和 PE 测序数据,同时支持 gzip 和 bzip2 压缩文件。
另外也支持 phred-33 和 phred-64 格式互相转化,现在之所以会出现 phred-33 和 phred-64 格式的困惑,都是 Illumina 公司的锅(damn you, Illumina!),不过现在绝大部分 Illumina 平台的产出数据也都转为使用 phred-33 格式了。
4. trimmomatic使用
- 简单使用
1
java-jar trimmomatic-0.30.jar PE -threads 20 -phred33 R1.fq R2.fq clean.R1.fq unpaired.R1.fq clean.R2.fq unpaired.R2.fq ILLUMINACLIP:TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:36
参数解释
|参数|解释|
|:—-|:—-|
|PE|选择测序数据的类型,是单端(SE)还是双端(PE)|
|-threads|线程数量,并发计算,提高效率|
|-phred33|一种数据格式,illumina测序数据的格式,也有-phred64,可以不选|
|ILLUMINACLIP:TruSeq3-PE.fa:2:30:10|切除adapter序列。参数后面分别接adapter序列的fasta文件:允许的最大mismatch数:palindrome模式下匹配碱基数阈值:simple模式下的匹配碱基数阈值。|
|LEADING|切除首端质量不足n的碱基(n由自己设定)|
|TRAILING|切除尾端质量不足n的碱基(n由自己设定)|
|SLIDINGWINDOW:4:15|滑窗修剪,一个Windows的size是4个碱基,其平均碱基质量小于15,则切除。|
|MINLEN|最小reads长度,筛去过短序列|
|CROP: |保留reads到指定的长度|
|HEADCROP: |在reads的首端切除指定的长度|
|TOPHRED33 |将碱基质量转换为pred33格式|
|TOPHRED64 |将碱基质量转换为pred64格式|
- 实操
nohup java -jar /B313/public_software/Quality_control/Trimmomatic-0.38/trimmomatic-0.38.jar PE -threads 40 a-BR-1_RRAS30349-V_1.fq a-BR-1_RRAS30349-V_2.fq clean_a-BR-1_RRAS30349-V_1.fq unpaired_a-BR-1_RRAS30349-V_1.fq clean_a-BR-1_RRAS30349-V_2.fq unpaired_a-BR-1_RRAS30349-V_2.fq ILLUMINACLIP:/B313/public_software/Quality_control/Trimmomatic-0.38/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:50 &
nohup java -jar /B313/public_software/Quality_control/Trimmomatic-0.38/trimmomatic-0.38.jar PE -threads 40 a-BR-1_RRAS30349-V_1.fq a-BR-1_RRAS30349-V_2.fq clean_a-BR-1_RRAS30349-V_1.fq unpaired_a-BR-1_RRAS30349-V_1.fq clean_a-BR-1_RRAS30349-V_2.fq unpaired_a-BR-1_RRAS30349-V_2.fq ILLUMINACLIP:/B313/public_software/Quality_control/Trimmomatic-0.38/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:110 &
日志输出
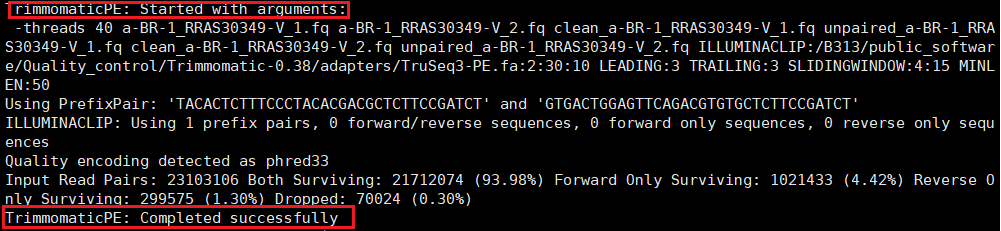
结果输出
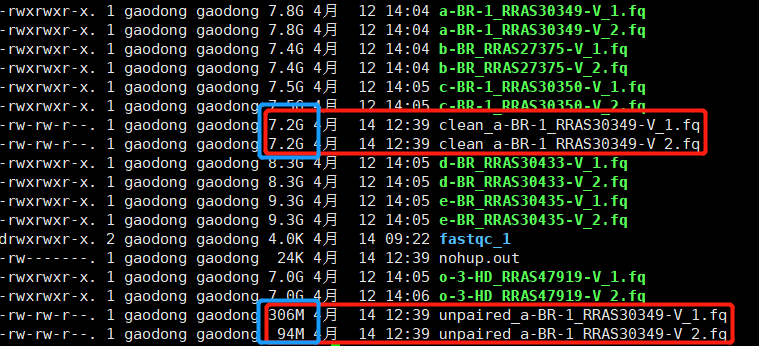
结果分析
由上图可以看出,最终输出的是4个文件,两个大文件是质控之后clean的数据,而另外两个小文件是被clean out的data,两者组合起来就是原来的数据。
剩余数据记录
nohup java -jar /B313/public_software/Quality_control/Trimmomatic-0.38/trimmomatic-0.38.jar PE -threads 40 b-BR_RRAS27375-V_1.fq b-BR_RRAS27375-V_2.fq clean_b-BR_RRAS27375-V_1.fq unpaired_b-BR_RRAS27375-V_1.fq clean_b-BR_RRAS27375-V_2.fq unpaired_b-BR_RRAS27375-V_2.fq ILLUMINACLIP:/B313/public_software/Quality_control/Trimmomatic-0.38/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:110 &
nohup java -jar /B313/public_software/Quality_control/Trimmomatic-0.38/trimmomatic-0.38.jar PE -threads 40 c-BR-1_RRAS30350-V_1.fq c-BR-1_RRAS30350-V_2.fq clean_c-BR-1_RRAS30350-V_1.fq unpaired_c-BR-1_RRAS30350-V_1.fq clean_c-BR-1_RRAS30350-V_2.fq unpaired_c-BR-1_RRAS30350-V_2.fq ILLUMINACLIP:/B313/public_software/Quality_control/Trimmomatic-0.38/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:110 &
nohup java -jar /B313/public_software/Quality_control/Trimmomatic-0.38/trimmomatic-0.38.jar PE -threads 40 d-BR_RRAS30433-V_1.fq d-BR_RRAS30433-V_2.fq clean_d-BR_RRAS30433-V_1.fq unpaired_d-BR_RRAS30433-V_1.fq clean_d-BR_RRAS30433-V_2.fq unpaired_d-BR_RRAS30433-V_2.fq ILLUMINACLIP:/B313/public_software/Quality_control/Trimmomatic-0.38/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:110 &
nohup java -jar /B313/public_software/Quality_control/Trimmomatic-0.38/trimmomatic-0.38.jar PE -threads 40 e-BR_RRAS30435-V_1.fq e-BR_RRAS30435-V_2.fq clean_e-BR_RRAS30435-V_1.fq unpaired_e-BR_RRAS30435-V_1.fq clean_e-BR_RRAS30435-V_2.fq unpaired_e-BR_RRAS30435-V_2.fq ILLUMINACLIP:/B313/public_software/Quality_control/Trimmomatic-0.38/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:110 &
nohup java -jar /B313/public_software/Quality_control/Trimmomatic-0.38/trimmomatic-0.38.jar PE -threads 40 o-3-HD_RRAS47919-V_1.fq o-3-HD_RRAS47919-V_2.fq clean_o-3-HD_RRAS47919-V_1.fq unpaired_o-3-HD_RRAS47919-V_1.fq clean_o-3-HD_RRAS47919-V_2.fq unpaired_o-3-HD_RRAS47919-V_2.fq ILLUMINACLIP:/B313/public_software/Quality_control/Trimmomatic-0.38/adapters/TruSeq3-PE.fa:2:30:10 LEADING:3 TRAILING:3 SLIDINGWINDOW:4:15 MINLEN:110 &
备注:在做完了质控之后要进行质量评价,先使用fastqc,然后使用multiqc进行整合,命令如下:
fastqc [-o output dir] [–(no)extract] [-f fastq|bam|sam]
[-c contaminant file] seqfile1 .. seqfileN
multiqc /B313/Zjunlin/raw_data/fastqc_1/ -o /B313/Zjunlin/raw_data/multi_qcraw/
5. 总结
Trimmomatic是一个专门的软件,用于进行质量控制,对于illumina的测序数据尤为有效。其实一般情况下我们是通过编程手动进行质控的,但是得益于广大生信开发工作者的努力,我们才有了这么方便的分析工具可以使用,让我们能够更加专注于生物数据的意义的分析,因此我们应该心怀感恩,和所有工作人员一起努力,为生物科学的发展贡献一份力量。
参考资料:
1. https://bio.biocoder.cn/book/15/content/5/
2. https://www.jianshu.com/p/21ad76e5577c
3. http://www.360doc.com/content/18/0111/12/19913717_721031558.shtml